Huixia Lu, estudiante becada por el China Scholarship Council (2016), defiende su tesis sobre el comportamiento de proteínas y pequeñas moléculas en el entorno de membranas celulares.
23/09/2020
La Huixia Lu defendió su tesis dirigida por Jordi Martí Rabassa. el 29 de septiembre de 2020 al Campus Nort de la UPC, titulada "Exploring free-energy landscapes and microscopic interactions of selected small-molecules and proteins with cell membranes”, la tesis describe las interacciones a nivel atómico entre sistemas moleculares de interés biológico y farmacéutico con los lípidos constitutivos de las membranas celulares.
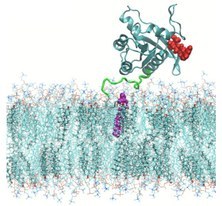
La presente Tesis doctoral está dedicada al estudio de las propiedades físico-químicas de moléculas pequeñas seleccionadas (por ejemplo aminoácidos como el triptófano u hormonas como la melatonina) y proteínas (como la KRas-4B) absorbidas en membranas celulares formadas por fosfolípidos y ubicadas en entornos fisiológicos. Dado que en estas condiciones, las membranas biológicas compuestas por fosfolípidos y colesterol están rodeadas de soluciones de electrólitos, entender las interacciones de la molécula pequeña o proteína con los fosfolípidos circundantes, el colesterol, el agua y todo tipo de especies iónicas es un tema de gran importancia fundamental. En particular, la presente Tesis se ha avanzado en el análisis de los aspectos estructurales y energéticos de una proteína oncogènica de la familia Ras, caracterizando las condiciones físicas que permiten que esta proteína se mantenga anclada a la célula. Las resultados descritos a la tesis pueden ayudar a dar luz a la comprensión de una gran variedad de cánceres, con un impacto directo en el diseño de medicamentos o tratamientos útiles para su curación.
Los lípidos considerados en esta Tesis incluyen los lípidossaturados dimiristoilfosfatidilcolina y dipalmitoilfosfatidilcolina, los lípidos insaturados dioleoilfosfatidilcolina y dioleoilfosfatidilserina y el colesterol. En esta Tesis se han aplicado simulaciones de dinámica molecular clásica y simulaciones de metadinámica bien temperada, de forma que todos los sistemas considerados han estado modelizados y simulados a nivel puramente atómico, con sistemas de hasta 200000 átomos.
Utilizando simulaciones clásicas de dinámica molecular (a escala 1 microsegundo), hemos estudiado la estructura y la dinámica microscópicas de las moléculas pequeñas y las proteínas KRas4B, estas últimas en formas pura y mutada (oncogénica). La membrana celular siempre se ha considerado en fase cristalina líquida, cosa que en algunos casos ha requerido aumentar la temperatura del sistema hasta 323 K. Se han calculado propiedades estructurales como por ejemplo el área por lípido y el grosor de la membrana, perfiles de densidad, parámetros de orden del deuterio, distribuciones orientacionals y el alcance de la penetración del agua a la membrana. Los coeficientes de difusión moleculares y las densidades espectrales atómicas revelan una gran variedad de escalas de tiempos que tienen un papel en la dinámica de membrana. El significado físico de todas las características espectrales de los lugares atómicos lipídicos se ha analizado y correlacionado con datos experimentales. El más relevante ha sido el hallazgo de la ubicación de lugares individuales de enlace de las varias sondas (pequeñas moléculas y proteínas) a la interfaz de la membrana. Finalmente, utilizando técnicas de trabajo reversible, se ha podido estimar la cantidad de energía libre necesaria para formar estos enlaces.
Mediante la aplicación de simulaciones de metadinámica bien temperada de 1 microsegundo, hemos realizado por primera vez cálculos de energía libre sistemática de la unión de las varias sondas a la membrana y al agua. Las superficies de energía libre muestran un comportamiento específico de los enlaces de moléculas pequeñas y proteínas a las membranas fosfolípidiques. Esta Tesis proporciona una metodología general para explorar superficies de energía libre en interfaces biológicas complejas que se pueden ampliar para estudiar otras interacciones de interés entre moléculas, péptidos, proteínas o fármacos y membranas en Química y Biología coloidal.
También hemos aplicado esta metodología para estudiar el caso de una proteína oncogènica prototípica (KRas), que se considera responsable de una gran variedad de cánceres. Nuestros resultados en superficies de energía libre indican la existencia de conexiones específicas de enlace de hidrógeno entre partes de la proteína (región hipervariabley cola farnesilada) que podrían ser responsables de la infección permanente de células sanas a través de su anclaje a la interfaz de la membrana.
Compartir: